里程碑!首次完整破译人类基因组
科学家首次破译完整人类基因组
3月31日,国际科学团队端粒到端粒联盟(T2T)发表第一个完整的、无间隙的人类基因组序列,首次揭示了高度相同的节段重复基因组区域及其在人类基因组中的变异,这是对2013年发布的参考基因组序列(GRCh38)的重大升级
图片来源:T。 POTAPOVA AND J。 GERTON/STOWERS INSTITUTE FOR MEDICAL RESEARCH
从1990年至今,我们一直在接近完整的人类基因组。今天,在发表于《科学》杂志的6项新研究中,科学家破解了最为关键的测序难题,获得了完整的人类基因组序列,或能将我们对人类的生长发育和疾病的了解,推向了新的高度。
撰文 | 栗子、clefable
人类的细胞里,通常有23对染色体。如果仔细观察这些染色体,你会发现组蛋白和缠绕在其上的DNA。我们的DNA由ATCG4种碱基排列而成,生命的密码就藏在其中。人类的基因组包含大约30亿个碱基对,排列极其复杂,但有规律,因为人与人基因组的相似性高达99.9%。多年来,科学家们一直想要借助其中的规律了解人体工作的原理,特别是与疾病相关的机制。
1986年,《科学》杂志上刊登了一篇由诺贝尔生理学或医学奖得主Renato Dulbecco撰写的文章。他认为,癌症研究已经来到一个关键的结点:要么零碎地挖掘一些关键的癌症基因,要么测定一个特定物种完整的基因组。而想要攻克人类的癌症,测定人的基因组,了解参与关键的生理学和病理学过程中各个基因是必不可少的。
同年,杜尔贝科和其他科学家联合发起了人类基因组计划(Human Genome Project)——对人类的核基因组进行完整地测序,1990年这项计划正式启动,被誉为生命科学领域的“登月计划”。又过了14年,也就是2004年,首个人类基因组测序结果发表,但基因组上仍有大约2亿个碱基未知,占全部序列的8%。这些未知序列当中包含很多高度重复的碱基序列,受限于当时的技术难以破译。
从那以后,科学家们陆续发布了越来越完整的人类基因组序列。到2017年,图谱上的缺口已经不足1000个,但人类基因组依然不够“完整”。
今天,一个由近100名研究人员组成的国际性的科学组织——端粒到端粒(T2T)联盟,在《科学》杂志上发表了6篇论文,表示他们测出了那些高度重复的DNA序列,并获得了迄今为止最完整的人类基因组T2T-CHM13,其中包括30.55亿个碱基对,由22条常染色体和X染色体无缝组装而成。此时,基因组的缺口仅剩5个,这项研究也被认为是首个完整的人类基因组测序。


突破难题
为了获得完整的基因组,团队首先要解决测序中的一个难点:人体内的大多数细胞都包含两个基因组——一个来自父亲,一个来自母亲。当研究人员将DNA片段组装起来时,父本和母本的序列会混合在一起,无法确定某一个基因组中实际发生的变异。
因此,研究团队使用了一个匿名的细胞系,这个细胞系来自20多年前从一位女性子宫里切除的异常生长物。那位女性经历了妊娠失败——精子进入一个没有染色体的卵细胞。如果受精卵中只有精子的遗传物质的话,就无法长成一个胚胎,但依然可以复制,尤其是在精子携带X染色体的时候。
异常的受精卵有个好处,就是只包含一个基因组,23对染色体都是两两相同的。华盛顿大学的遗传学家、协助领导人类基因组计划的罗伯特·沃特斯顿(Robert Waterston)说,这对填补基因组中的缺口有很大的帮助,因为测序仪不再需要解决父母染色体不一样的问题了。
除此之外,还有一个更重要的难点,就是高度重复的序列。在进行基因组测序时,科学家通常需要将DNA切成较短的片段再逐一测序,然后将测序结果拼凑起来。但遇到大量高度相似的序列时,研究者便难以确定它们之间的排列顺序。因此,科学家需要借助更加先进的技术,每次测量更长的DNA序列,来减少拼凑的需求。
于是,T2T联盟的科学家使用了多项前沿的测序技术,包括读取10万个碱基的纳米孔测序设备(nanopore device)和一个更加精确但每次只能读取约1万个碱基的测序仪等。团队把这些手段用在一起,几乎消灭了所有的基因组难题,只剩下5个缺口,总共大约1000万个未知碱基。另外,由于那个细胞系中只有常染色体与X染色体,Y染色体的碱基序列还需要详细解析。
首次确定的序列
此次,研究团队为基因组填补了大约2亿个碱基,主要位于着丝粒区域和近着丝粒染色体的短臂区域,还有许多长度超过1000个碱基的大段重复序列。这些序列中包含1900 多个基因,大部分是已知基因的拷贝,但也包括182个新基因。
研究团队在着丝粒上发现了一段特别的序列,它可以和一种名叫“动粒”的蛋白复合物结合,而“动粒”参与调控着染色体的移动,也参与染色体的分裂过程。一旦这个序列发生变异,就可能影响细胞内的基因表达,从而导致癌症。

4种类型的染色体,其中从左到右地3个为近着丝粒染色体。这类染色体的短臂非常短。图片来源于Vedantu
科学家也发现,着丝粒区域出现了异常高的变异水平。他们分析,着丝粒以及附近区域呈现出“分层扩散”的演化模式,即存在着不断重复、但有突变的序列,新的序列会和旧序列连接,而旧的序列被新的序列挤到边缘,慢慢缩短。
除此之外,5条近着丝粒染色体的短臂区域的测序结果显示,这些短臂区域中包含了编码核糖体RNA的基因的多个拷贝,一共400份。更重要的是,短臂区域的变异水平也比较高,并有大量的重复序列,如可移动的转座子等。研究团队认为,短臂区域很可能是基因演化的热点区域,因为停留在那里的基因拷贝可以自由变异并获得新的功能。

图中右侧为染色体,它含有两条姐妹染色单体,它们的连接处为着丝粒。图片来源:National Human Genome Research Institute
科学家们十分重视基因组中的重复序列,并给它们编排了目录。在这些序列当中,碱基上的化学修饰可能和许多疾病有关,例如一些神经障碍和发育障碍等。并且那些疾病的出现,通常和某些重复序列的拷贝数量变化有关。
后续的计划
毫无疑问,这个新的人类基因参考图谱将会对基因组分析产生重大影响。它将能更好地支持个性化医疗、人群基因组分析和基因组编辑。欧洲分子生物学实验室的副主任、生物信息学家尤恩·伯尼(参与了人类基因组计划,没有参与这些研究)表示:“即使是5年前,我们也没有想到能做到这一点,更不用说10年前了。这些研究工作极为出色。”基因编辑公司Inscripta的基因组学家迪安娜·丘奇(Deanna Church)认为,这些此前未知的DNA区域让我们对基因组有了更多新的了解。
虽然,这些新的研究工作使基因组测序达到了一个新的里程碑,但人类基因组测序仪器并没有完全下班。由于使用的细胞系没有Y染色体,T2T联盟的科学家获取了哈佛大学系统生物学家列昂尼德·佩什金(Leonid Peshkin)的基因组,并对他的一条Y染色体进行了测序。此前,佩什金博士的基因组已经被研究得相对充分了,只是还没有得到完整的基因组序列。好消息是,昨日,T2T联盟在推特上宣布他们已经确定了Y染色体上缺失序列的正确排列形式。想必离解析出完整的Y染色序列也不远了。
除此之外,人类基因组计划联合负责人、贝勒医学院遗传学家理查德·吉布斯(Richard Gibbs)说:“还有一些工作要做。”包括他在内的许多研究人员都强调,现在需要从更多样化的人群中获取完整的基因组序列,来寻找染色体短臂中可能存在的变异,以及其他获取其他测序难度比较高的区域的变异,因为这些都有可能和疾病或别的性状有关。
此外,他们还有一个新目标——从不同种族或血统的人中提取350个基因组(目前已破译了70个基因组)。这些基因组是人类泛基因组参考联盟(Human Pangenome Reference Consortium)的一部分,它们的测序更有挑战性,因为每一对染色体都是两两不同的。最终,科学家希望给每一个基因组,都找出端粒到端粒的完整序列。
参考链接:
https://www.science.org/content/article/most-complete-human-genome-yet-reveals-previously-indecipherable-dna
https://www.science.org/toc/science/current
https://www.science.org/doi/10.1126/science.abj5089
https://www.science.org/doi/10.1126/science.abk3112
https://www.science.org/doi/10.1126/science.abl4178
https://www.science.org/doi/10.1126/science.abo5367
https://www.science.org/doi/10.1126/science.abj5089
本文转自《环球科学》
柳叶刀:新冠变异株可能正在免疫低下感染者体内
近日,国际权威医学期刊《柳叶刀》发表了一篇题为《下一个新冠病毒“关切变异株(VOC)”在哪》(WhereisthenextSARS-CoV-2variantofconcern?)的评论。评论表示,某个免疫力低下的新冠感染者体内可能正在进化着一个新冠病毒变体:它具有高度传染性、能挑战现有免疫和疗法。0000这就是Intel显卡的意义!Arc A750限量版199美元史低价达成
NVIDIA已经推出了RTX4060Ti,定价399美元(3199元人民币)。AMD马上推出RX7600,定价269美元(国行稍后告诉大家)。红绿两家针锋相对之时,蓝色阵营的IntelArc显卡也来凑热闹了,ArcA750限量版杀到了199美元,A7系列史上首次跌破200美元大关。诺哈网2023-05-26 03:50:010000诞生于千年之前心理学理论,拿还珠格格解释你就懂了!
来源:原点阅读题记:知道是什么样的人得病比知道一个人得的是什么病更为重要。——希波克拉底有很多我们以为历史很长的事物,其实是最近几十年才有的。比如说现在遍布城市每个角落的新疆美食大盘鸡,其实是20世纪80年代后期才有的;比如说我们旅行的时候都会用到的拉杆箱,其实出现于1991年,距今还不到30年;再比如说我们现代人都知道大脑是人用来思考的器官,但是在古代却并非如此。诺哈网2023-05-26 03:49:560000蘑菇也会说话,还能说50个词!不,它可能只是在吧唧嘴!
世界上并不是只有人类才会说话,很多生命都会互相交流环境信息,比如哪儿有吃的,哪儿有危险,能够趋利避害。科学家就曾发现蜜蜂通过跳舞来告诉同伴哪儿有上好的花蜜资源,做出相关研究的科学家还获得过1973年的诺贝尔生理学医学奖。植物也并非傻乎乎地呆在原地,它们会释放挥发性有机化合物,来吸引传粉者、吓退天敌,或者向临近植株传递环境中的危险信息。诺哈网2023-05-25 23:08:550000中国科学家再次实现二氧化碳“变废为宝”:还原合成葡萄糖和油脂
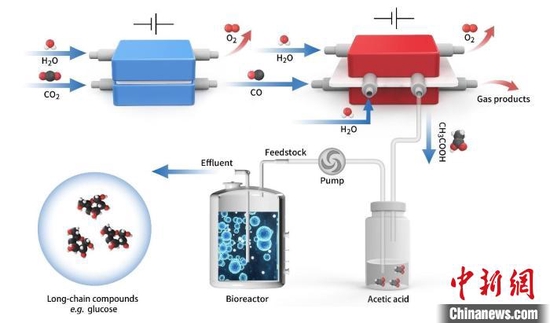
体外二氧化碳人工合成高能长链食品分子示意图。研究团队供图中新网北京4月28日电(记者孙自法)二氧化碳除了可以“变”淀粉,还能“变”其他东西吗?中国科学家最新给出的答案是“能”——可以还原合成葡萄糖和油脂。诺哈网2023-05-25 20:42:500000